A la suite de mon intervention pour Podcast Science où je discutais des virus, je suis tombé sur cette citation d’Henri Michaux dans son recueil de poèmes “Face aux verrous”:
Tout virus est prodigue
Cela résume bien la fascination qu’on peut avoir pour ces fantastiques entités qui brisent nos barrières immunitaires mais aussi nos conceptions, nos définitions et les idées reçues. Rentrons donc ensemble dans le fascinant monde viral.

Une question qu’on pose souvent concernant les virus, c’est celle de leur position dans l’arbre du vivant. D’emblée, ça semble une tâche bien ardue puisque la plupart des chercheurs ne sont pas d’accord pour considérer que les virus sont des organismes vivants. C’est une prudence qui peut se comprendre : vu qu’on se casse déjà le cul à tenter de classer le vivant (1, 2 & 3), autant ne pas y rajouter des trucs qui n’ont rien à voir comme des silex, des trombones ou le sécateur de votre voisin.
Mais alors pourquoi les virus ne seraient-ils pas vivants ? Pour comprendre l’origine de la controverse, il faut s’intéresser tout d’abord aux premières définitions de ce qu’on a appelé virus. En 1957, André Lwoff, célèbre virologue, écrit un article où il tente de retracer l’histoire de sa discipline et celle du concept du virus. Il commence son article en rappelant tout d’abord l’étymologie du mot virus qui vient donc du latin et signifie ‘toxine, poison’. Puis il évoque le fait qu’après les découvertes de Louis Pasteur, tout agent microbien qui entraînait des maladies infectieuses était appelé virus, même s’il s’agissait de bactéries, de champignons ou d’autres organismes unicellulaires. En gros, tout ce qui nous faisait tomber malade, et qu’on ne voyait pas à l’œil nu, était un virus… Pas génial pour réaliser des classifications cette définition... Puis on a commencé à distinguer les agents infectieux qu’on pouvait voir avec de forts microscopes de ceux qui restaient totalement invisibles, semblant passer entre les mailles des filets des chercheurs qui les étudiaient. Quand je parle de maille, ce n’est pas entièrement rhétorique : à l’époque, pour stériliser certaines solutions, on les faisait passer à travers un gel composé de fibres de polymères qui forment des mailles de 200 nm (milliardième de mètre). A l'époque, on ne connaissait aucune bactérie qui soit assez petite pour traverser ces mailles. Si des agents infectieux passaient à travers ces mailles, on avait donc tendance à penser qu’ils étaient trop petits pour constituer des organismes vivants. Le filtrat était plutôt considéré comme un ‘fluide infectieux’. A partir de là, seuls ces agents infectieux furent appelés « virus ».
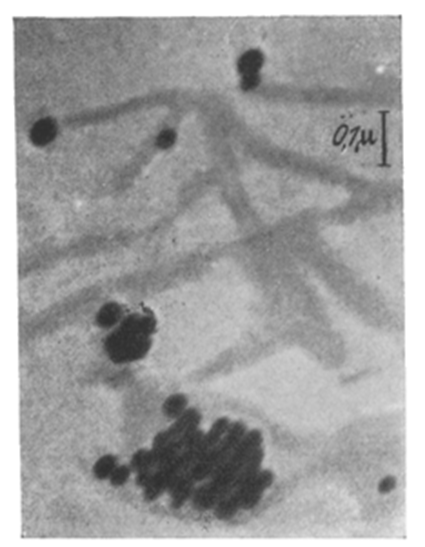
Mais très vite des chercheurs identifient, à l’aide de puissants microscopes électroniques, de minuscules particules dans le fluide infectieux et associent l’infection avec ces particules : du coup ce sont ces particules qu’on appellera virus. On leur découvre certaines spécificités : l’incapacité de réaliser une division binaire comme le font les bactéries (donc de se répliquer de manière autonome), la présence d’un matériel génétique composé d’ADN ou d’ARN codant un nombre restreint de protéines (alors que tous les autres vivants possèdent de l’ADN et de l’ARN, jamais l’un sans l’autre). Enfin, contrairement à tout organisme vivant cellulaire, les virus semblent incapables de capturer et de stocker de l’énergie, c’est-à-dire d’avoir un métabolisme.
Pour résumer, les virus, ce sont de toutes petites particules, avec la moitié de ce qu’il faut pour avoir un véritable matériel génétique et ce sont des parasites obligatoires.
Mais ça, j’ai envie de dire, c’était avant. Car en biologie, on découvre souvent que la nature s’en contrefout de nos définitions…
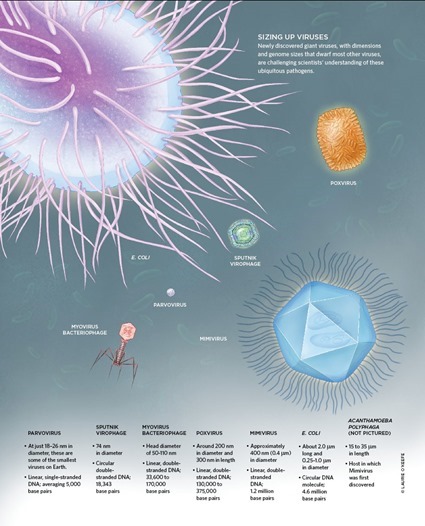
Déjà niveau taille, la définition du minuscule virus ne tient plus depuis le début du XXIème siècle. En effet, en 2003 parait la description de Mimivirus, un virus hors du commun : isolé en 1992 à Bradford, en Angleterre, dans une tour d’eau de refroidissement, Mimivirus fut initialement considéré comme une toute petite bactérie (surnommée Bradford coccus).
Pourquoi? Parce qu’il mesurait 400nm de diamètre (2 fois plus grand que les mailles stérilisantes, vous vous souvenez ?). Pourtant, impossible de cultiver cette prétendue minibactérie en dehors de l’organisme dans lequel on l’avait trouvé, l’amibe Acanthamoeba polyphaga. Une autre manière de caractériser une bactérie, c’est d’isoler et séquencer les gènes qui leur permettent de synthétiser des protéines : les gènes ribosomiques. Mais là encore, c’est chou blanc : impossible de trouver des gènes ribosomiques dans le parasite mystérieux de l’amibe. Finalement, cet exemple nous montre la limite et surtout la puissance des définitions : en effet, dans la tête des chercheurs qui ont découvert cet étrange organisme, la taille des virus ne pouvait pas dépasser les 200nm. Il a fallu plus de 10 ans avant qu’une autre équipe de chercheurs se mettent en tête de caractériser l’organisme mystérieux en séquençant intégralement son génome. Et là, plus de doute possible, il s’agit bien d’un virus qu’ils vont appeler Mimivirus (pour Mimicking microbe virus – le virus imitant les microbes).
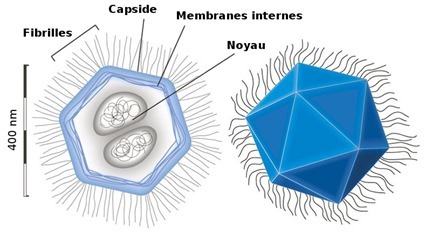
Son génome semblait gigantesque à l’époque puisqu’il atteignait la taille de 800 000 nucléotide ou 800kpb (c’est-à-dire 800 000 lettres A, T, G, C qui permettent de coder les gènes du Mimivirus). Le record était avant détenu par un virus bactérien dont le génome atteignait 670kpb alors que les génomes bactériens les plus petits peuvent tomber à 580 kpb.
Depuis cette découverte, c’est la chasse aux virus géants : en 2009, les marseillevirus…
… en 2010 Megavirus chilensis
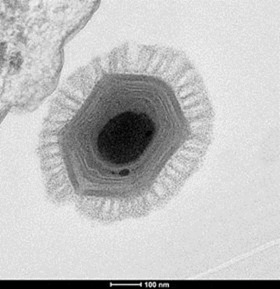
… tous ces virus appartenant au groupe des Megaviridae caractérisés par une enveloppe icosaédrique.

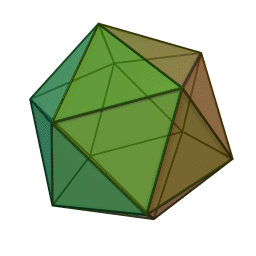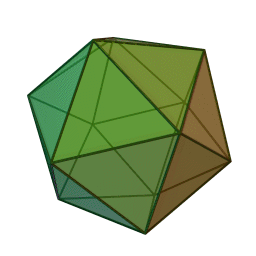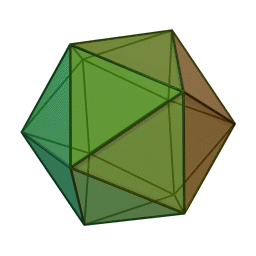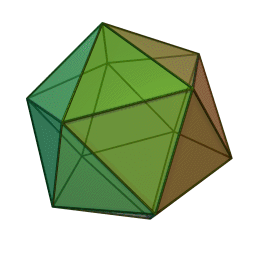
A chaque découverte, c’est aussi un record mondial du génome le plus grand, qui plafonne aujourd’hui à 1,26 Mb, c’est-à-dire 1,25 fois plus grand que le premier mimivirus découvert.
Puis en 2013, découverte du Pandoravirus dont l’enveloppe, cette fois-ci, ressemble à une amphore. Leur taille est gigantesque pour des virus : 1,2 µm de long et leur génome atteint 2,8 Mb. Il s’agit bel et bien d’une nouvelle famille car ni leur forme, ni leur génome ne ressemble à celui des Megaviridae. Et comme on n’a pas fini de rigoler, une équipe vient de découvrir en 2014, congelé dans le permafrost sibérien, un virus en forme d’amphore mais dont le génome ressemble à celui des Megaviridae…
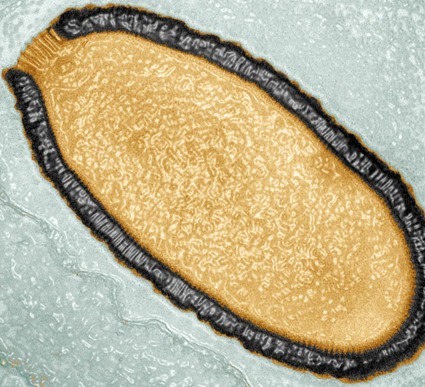
C’est quoi ce virus ? Un hybride de Pandoravirus et Megavirus ? Megapandoravirus ? Sobrement, leur découvreur l’ont nommé Pithovirus puisque pithos signifie amphore en grec, même si perso j’aurais choisi foutlamerdevirus. J’imagine que ça a pu tenter certains de ces chercheurs, notamment au vu de la réaction du principal auteur de l’étude qui a dit, je cite, ‘je ne comprends plus rien’…
La découverte est un exploit en soi puisqu’ils ont quand même caractérisé un virus qui était resté emprisonné dans la glace pendant près de 30000 ans… mais qui a conservé sa capacité virulente ! (oui, ils ont testé, parce que le monde n’est pas assez effrayant au goût de certains). Vous inquiétez pas trop quand même : Pithovirus est spécifique d’une espèce d’amibe et probablement inoffensif pour les humains… [Mais si vous insistez pour vous foutre les chocottes, rien ne nous dit que dans le sillage du réchauffement climatique, des virus mortels pour les humains ne puissent pas accompagner la décongélation des glaciers… lolilol]
Mais au lieu de déprimer, ébahissons-nous un peu sur la perspective que nous ouvrent ces nouveaux virus qui malmènent les définitions. En effet, ils sont grands, avec des génomes très conséquents. Du coup certains chercheurs se demandent si cette grande taille et ces grands génomes ne seraient pas des caractères ancestraux, perdus chez la majorité des petits virus que l’on connaît mieux. Considérer cela ouvre la possibilité de les inclure dans l’arbre du vivant.
Si vous vous souvenez de mes trois articles sur l’arbre du vivant (1, 2 & 3), vous vous rappellerez peut-être que depuis les travaux de Woese, Kandler et Wheelis, l’arbre du vivant est représenté par trois branches, trois domaines de la vie, celui des Archées, des Eubactéries et des Eucaryotes.
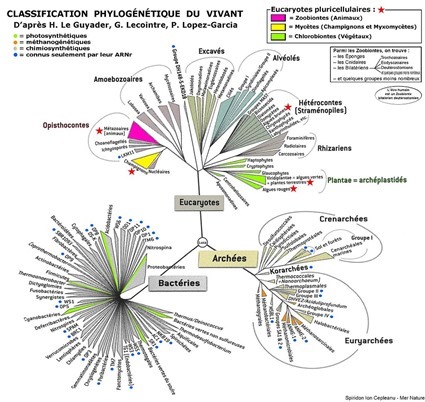
Quand on travaille sur la définition des relations de parenté entre des organismes si différents, trouver des caractères permettant des comparaisons est un peu compliqué. Par exemple la comparaison d’une trompe d’éléphant, de la queue d’une cerise et du flagelle d’une bactérie est assez vide de sens…


Heureusement, il existe un caractère partagé par l’ensemble des trois domaines et qui permet de nombreuses comparaisons: c’est l’ADN. En effet, tous ces organismes possèdent un matériel génétique fait d’ADN qui contient des informations pour construire des protéines et dont le code est, à quelques rares exceptions près, universel. Chic : ça veut dire qu’on peut comparer, dans les 3 domaines du vivant, différents gènes, qui codent des protéines qui vont nous permettre d’évaluer qui est plus proche de qui en termes de parenté. Le problème, c’est qu’il faut comparer des protéines qui ont été conservées chez ces trois domaines durant l’évolution (des protéines ancestrales donc). Historiquement, les gènes utilisés pour cette comparaison étaient ceux qui donnent des ribosomes: chaque domaine du vivant possèdent des ribosomes (et donc on peut envisager que leur ancêtre avait des ribosomes). Les ribosomes, ce sont les petites usines cellulaires qui permettent de lire des molécules d’ARN pour synthétiser une protéine. Pour rappel, pour ces 3 domaines du vivant, le schéma de fonctionnement classique pour arriver à la confection de protéine passe de l’ADN à l’ARN par une étape de transcription, et de l’ARN à la protéine par une étape de traduction.
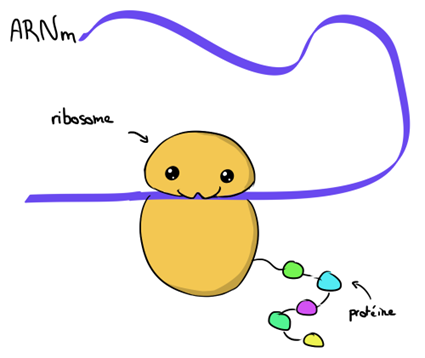
Revenons-en à nos fameux gènes ribosomiques: grâce à la comparaison de ces gènes, on a découvert que les organismes cellulaires pouvaient être classés en trois domaines distincts correspondant à trois lignées évolutives distinctes. Ces lignées sont tout de même apparentées (elles ont par exemple conservé le caractère ancestral d’un matériel génétique avec son code plus ou moins universel) et sont donc les descendantes d’un ancêtre commun putatif qu’on appelle classiquement LUCA (pour Last Universal Common Ancestor - dernier ancêtre commun universel).
Et les virus? Ben là on est un peu dans le caca. Si on veut comparer la situation avec les 3 autres domaines, il faut obtenir leur séquence d’un gène ribosomique, et vous me voyez venir: les virus n’ont pas de gènes ribosomiques… En même temps, ce sont des parasites obligatoires qui dérivent la machinerie cellulaire de leur hôte bactérien, archéen ou eucaryote pour justement se passer d’avoir des ribosomes viraux. A l’instar du ‘pas de bras, pas de chocolat’, les virus se sont vus dire ‘pas de ribosomes, pas de domaine du vivant’.
Mais y’a ce matériel génétique tout de même: c’est quand même dingue que les virus utilisent un matériel avec le même code universel, non? On peut raisonnablement se permettre d'envisager un scénario évolutif dans lequel les virus possédaient, il y a très longtemps, des gènes codant pour des ribosomes, mais qu’au hasard des mutations génétiques, ils se soient vus privés de la capacité de les utiliser, les poussant dans une course évolutive à devenir les parasites les plus efficaces qui soient. Alternativement, il existe d’autres scénarios qui peuvent expliquer la présence de ce matériel génétique et l’absence de ribosomes. A l’instar des cellules cancéreuses qui, spontanément, sont générées par les organismes multicellulaires, les virus pourraient être les rejets des 3 domaines du vivant. Vu que les virus sont super diversifiés, cela pourrait simplement être le résultat de ces vomis parasitiques du vivant à différents moments de l’évolution… Le vivant aurait généré sa propre arme de destruction massive au final.
Rentrer dans le détail serait un poil trop compliqué. Cependant, je vais vous présenter des résultats récents concernant l’origine des virus. Quand je vous ai dit que pour réaliser des arbres de parenté, on utilisait des gènes ribosomiques, je vous ai parlé d’une méthode classique et simple. Mais de nombreux chercheurs utilisent d’autres gènes pour classer les organismes car dans certains cas, les gènes ribosomiques ne suffisent pas à résoudre des nœuds de parenté. Après la découverte des Mimivirus, certains chercheurs ont décidé d’utiliser les séquences de ces génomes de virus géants pour voir où ces gènes pouvaient placer les Mimivirus dans l’arbre du vivant. En étudiant leur génome, ils furent déjà pas mal surpris de trouver des gènes codant des protéines intervenant dans la synthèse de protéines. Certes pas des ribosomes, mais des gènes accessoires à la formation des protéines. Chez les mimivirus, ce groupe de gènes mène à la formation de petites usines virales qui permettent de répliquer le virus sans utiliser de ribosomes. Encore une fois, on peut se demander s’il s’agit là d’un reliquat ancestral d’un répertoire de gènes dédié à la synthèse de protéine, ou s’il s’agit plutôt d’un vol de gène d’origine bactérienne, archéenne ou eucaryote. Mais le génome des Mimivirus contient aussi des gènes qui permettent la formation de petites usines à convertir l’ADN en ARN, ce qu’on appelle les ARN polymérases. Or, en utilisant les séquences de ces ARN polymérases pour les comparer entre les mimivirus et entre les 3 domaines du vivant, des chercheurs se sont aperçus que les arbres de parenté obtenus permettaient la classification des mimivirus en un 4ème domaine du vivant. Tout fous, ils se sont dit qu’ils allaient réaliser des arbres systématiquement pour tous les gènes contenus dans le génome des mimivirus…
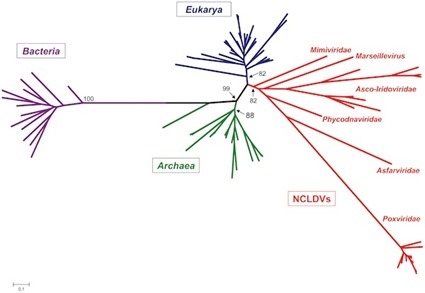
Et là, c’est le drâme… Enfin, c’est un bordel sans nom… Des gènes de mimivirus se groupent tantôt tout seuls, tantôt chez les eucaryotes, les eubactéries ou les archées. Selon certains, ces résultats montrent que les virus, et les mimivirus en particuliers, sont les rois des voleurs de gènes. Le problème c’est que ces vols de gènes, ce qu’on appelle couramment des transferts horizontaux de gènes, laissent des traces qui s’effacent au fur et à mesure de l’évolution. Même si les virus évoluent vite, force est de constater que la plupart de ces transferts sont probablement très très vieux (on trouve des transferts horizontaux récents dans leurs génomes, mais ils sont facilement reconnaissables).
Alternativement, d’autres chercheurs proposent un autre angle d’approche pour expliquer ce bordel génétique: l’hybridation, et les transferts génétiques. Comme on l’a vu lors de l’interview de Marc-André Sélosse, les transferts de gènes et autres cochoncetés génétiques sont légions entre les 3 domaines du vivant. Du coup, face à la figure classique de l’arbre du vivant, ces chercheurs évoquent maintenant la possibilité de représenter les relations de parenté où les branches peuvent s’anastomoser comme un système de racine, ou comme les bras des cours d’eau près d’un delta : une sorte de rhizome du vivant, pour utiliser l’expression de Didier Raoult qui est le premier à avoir proposé ce scénario…

C’est vrai que classer des domaines du vivant quand ceux-ci peuvent échanger des gènes facilement, c’est un peu comme se demander « qui est plus proche de qui » à des personnes qui seraient en mesure de s’échanger à volonté des organes: si j’ai le cerveau de Robert, les reins de Jocelyne et le cœur de Jean-Louis, est-ce que je suis encore « moi-même » ? Et quelles sont mes relations de parenté avec les familles des donneurs ? La nature nous aurait concocté au grès des milliards d’années d’évolution, des mini-frankenstein avec lesquels elle nous laisserait nous dépatouiller pour en déterminer la provenance. Les 4 domaines du vivant, virus inclus, seraient au final des mosaïques génétiques.
Si vous vous souvenez de l’émission avec le Prof. Sélosse, vous vous rappellerez que les eucaryotes sont constitués en partie d’eubactéries qui leur permettent de respirer ou de faire de la photosynthèse : les mitochondries et les chloroplastes. Les eucaryotes ont aussi beaucoup de gènes en commun troqués avec des virus, des archées ou des eubactéries. Un joyeux bordel génétique… qui permet l’émergence de théories assez extravagante. Il y a par exemple ce ptit scénario évolutif bien controversé (le scénario évolutif en mode troll) : une des caractéristiques des eucaryotes est de posséder un noyau qui contient leur matériel génétique. Et si ce noyau était une contribution virale à l’évolution des eucaryotes ? [insérer musique d'Inception]
Bon allez, quittons le mode troll… en fait non, on va plutôt essayer de vous faire considérer les virus d’un œil sympathique tiens! Mission impossible? On va bien voir…
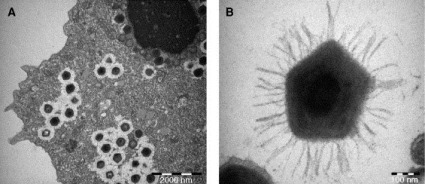
Commençons par les Mimivirus: Ces virus sont tellement grands qu’ils sont les premiers chez qui on a découvert qu’ils pouvaient s’offrir le luxe d’être parasités… par des virus.
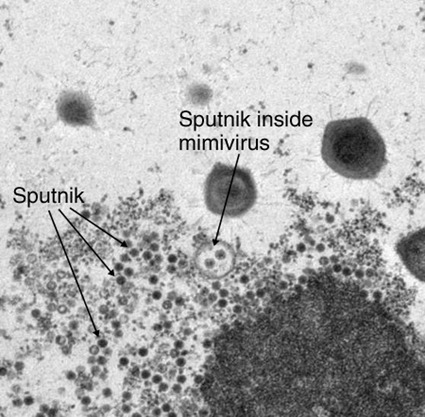
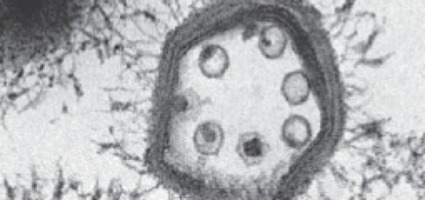
Ces virus de virus, ou virophages, ont été découverts en 2006 par microscopie électronique dans les recoins d’un mimivirus appellé mamavirus (les virologues sont les rois du calembour). Le premier virophage a été appelé Sputnik, et possède un tout petit génome de 18000 pb qui utilise le matériel viral du mamavirus pour se répliquer. On a donc ici, un virus de virus d’une amibe (qui peut être dans certain cas un parasite – c’est donc un parasite de parasite de parasite). Ça vaut bien quelques applaudissements, non ? Et si je vous disais que certains virus apportaient un avantage non négligeable à l’hôte qui le porte, au lieu de lui porter détriment?
De nombreuses guêpes parasitaires sont ainsi entrées en symbiose avec des virus dont elles sont les hôtes. C’est le cas de la guêpe Cotesia glomerata qui parasite les chenilles de la piéride du chou qui en temps normal se métamorphosent en papillon blanc et noir.

Si Cotesia arrive à choper une chenille, elle lui pond ses œufs à l’intérieur de son corps qui va alors servir de garde-manger mobile pour les larves jusqu’au moment de leur métamorphose.
Mais au cours des milliers de générations de parasitismes, les chenilles ont développé un système immunitaire leur permettant de lutter contre l’invasion de larves de guêpe dans leur corps. Mais c’est sans compter les virus des guêpes qui vont déprimer le système immunitaire de la chenille.
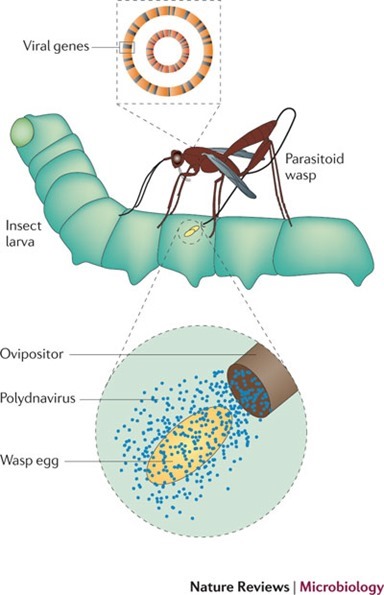
C’est comme si ces guêpes étaient rentrées dans l’ère de l’armement biologique avec un virus comme moyen de destruction massive du système immunitaire. Certains chercheurs pensent même que plusieurs gènes portés par le virus et qui servent à perturber le système immunitaire de la chenille, voire son comportement, auraient pour origine… le génome de la guêpe. Cela revient à ce que la guêpe emploie un virus pour exprimer ses propres gènes dans la chenille pour faciliter son parasitisme. Virus for the win!
Dans un autre cas, la guêpe Aphidius ervi parasitant les pucerons (et utilisée par les humains comme pesticide biologique), réussit à parasiter les pucerons malgré leurs défenses immunitaires.

Ces défenses immunitaires de pucerons sont assistées par une flore bactérienne essentiellement composée de la bactérie Hamiltonella defensa dont le potentiel néfaste contre les larves de guêpes est effectif grâce à un virus spécifique de la bactérie qui produit une toxine ciblant les larves de guêpes. Comme quoi les virus ne sont pas systématiquement du côté des guêpes.
Au-delà de leur rôle surprenant dans des interactions entre espèces, les virus sont probablement à l’origine de changements évolutifs étourdissants tant ils ont permis de façonner les espèces actuelles.
Marc-André Sélosse nous expliquait par exemple que chez différents mammifères comme les humains et les primates, une partie du placenta est formée grâce à une protéine d’origine virale appelée syncytine. A l’origine cette protéine permet à certains virus comme celui de la grippe de fusionner leur propre enveloppe aux cellules pour pouvoir les infecter. Par un incident génétique, ce gène s’est retrouvé inséré dans le génome de primates et a permis d’assister la formation d’une partie du placenta qui nécessite la fusion entre les tissus du fœtus et celui de l’utérus de la mère.
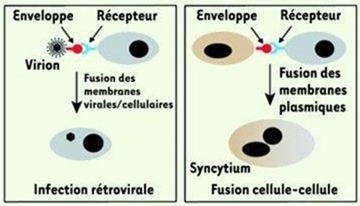
C’est sympa comme contribution virale, non?
A vrai dire, l’insertion de gènes de virus dans le génome de son hôte n’est pas si accidentelle que ça. On entend souvent parler de virus latents : il ne faut cependant pas l’imaginer en train de feuilleter gentiment un Paris-Match dans un recoin de la cellule en attendant un coup de stress. Cette latence correspond le plus souvent à une insertion du génome du virus dans le génome de la cellule infectée. Mais parfois cette latence peut sauter des générations! C’est dans le cas où le virus s’est intégré dans le génome de cellules à l’origine de gamètes sexuels (ovules ou spermatozoïdes).
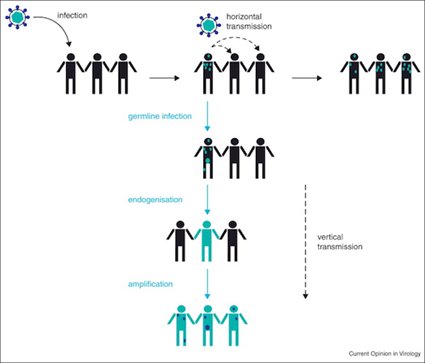
Dans ce cas-là, le génome du virus peut être transmis de génération en génération (on parle de transmission verticale, suivant le sens généalogique, contrairement à la transmission horizontale qu’on refile à son voisin en lui éternuant dans la tronche). Le phénomène n’est pas si rare qu’on le croie et on peut par exemple dénombrer près de 100000 gènes d’origine rétrovirale dans le génome humain (les rétrovirus étant les champions pour la latence). Par comparaison, on n’a que 20000 gènes à protéines dans le génome. Ces gènes sont pour la plupart inactivés, mais peuvent, dans des cas particuliers, redevenir mobiles et sont ainsi probablement à l’origine de la diversité génétique de notre groupe, mais aussi d’autres organismes comme les plantes, des insectes, etc. Sans ces virus, nous ne serions probablement pas si complexes, la vie ne serait pas si plastique et changeante, l’évolution n’aurait probablement pas eu tant de matériau pour façonner ces formes construites avec raffinement, si différentes les unes des autres, et dépendant les uns des autres d'une manière si complexe…
Cette phrase vous rappelle quelque chose? C’est en réalité la dernière phrase de l’origine des espèces de Charles Darwin… une citation hackée par un virus.
Liens:
Viruses Reconsidered
Our Inner viruses 40 million years in the making
Viruses as a cure
Gigantic Giant Viruses and the endless viral frontier
Meet your new symbionts: several trillion viruses
Giant viruse resurrected from 30000 year old ice
We are viral from the beginning
Koala pandemic genetics
Russian doll warfare virus bacteria aphid wasp
Making viruses the natural way
Mammals made by viruses
Please welcome megavirus
Glimpse of the fourth domain
Dark matter hints at fourth domain of life
Genomes of giant viruses hint at fourth domain of life
Trouble in the fourth domain
Références :
Albers, S. V., P. Forterre, D. Prangishvili and C. Schleper (2013). "The legacy of Carl Woese and Wolfram Zillig: from phylogeny to landmark discoveries." Nat Rev Microbiol 11(10): 713-719.
Boughalmi, M., P. Colson and B. La Scola (2013). "Mimiviruses and Marseilleviruses, the Largest Known Viruses." Encyclopedia of Life Sciences.
Boyer, M., S. Azza, L. Barrassi, T. Klose, A. Campocasso, I. Pagnier, G. Fournous, A. Borg, C. Robert, X. Zhang, C. Desnues, B. Henrissat, M. G. Rossmann, B. La Scola and D. Raoult (2011). "Mimivirus shows dramatic genome reduction after intraamoebal culture." Proc Natl Acad Sci U S A 108(25): 10296-10301.
Boyer, M., M. A. Madoui, G. Gimenez, B. La Scola and D. Raoult (2010). "Phylogenetic and phyletic studies of informational genes in genomes highlight existence of a 4 domain of life including giant viruses." PLoS One 5(12): e15530.
Cann, A. J. (2015). "Virus Structure." Encyclopedia of Life Sciences: 1-9.
Colson, P., G. Gimenez, M. Boyer, G. Fournous and D. Raoult (2011). "The giant Cafeteria roenbergensis virus that infects a widespread marine phagocytic protist is a new member of the fourth domain of Life." PLoS One 6(4): e18935.
Domingo, E. and C. Perales (2014). "Virus Evolution." Encyclopedia of Life Sciences.
Fauquet, C. M. and G. P. Martelli (2013). "Viral Classification and Nomenclature." Encyclopedia of Life Sciences.
Forterre, P. (2006). "The origin of viruses and their possible roles in major evolutionary transitions." Virus Res 117(1): 5-16.
Forterre, P. (2013). "The virocell concept and environmental microbiology." ISME J 7(2): 233-236.
Forterre, P., M. Krupovic and D. Prangishvili (2014). "Cellular domains and viral lineages." Trends Microbiol 22(10): 554-558.
Gaia, M., P. Colson, C. Desnues and B. La Scola (2013). "Virophage Concept, The." Encyclopedia of Life Sciences.
Georgiades, K., V. Merhej and D. Raoult (2011). "The influence of rickettsiologists on post-modern microbiology." Front Cell Infect Microbiol 1: 8.
Haig, D. (2012). "Retroviruses and the placenta." Curr Biol 22(15): R609-613.
Holmes, E. C. (2014). "Freezing viruses in time." Proc Natl Acad Sci U S A 111(47): 16643-16644.
La Scola, B., S. Audic, C. Robert, L. Jungang, X. de Lamballerie, M. Drancourt, R. Birtles, J. M. Claverie and D. Raoult (2003). "A giant virus in amoebae." Science 299(5615): 2033.
Landires, I. (2010). "The post-Darwinist concept of species: a place for Lamarck?" Lancet 375(9717): 806.
Legendre, M., J. Bartoli, L. Shmakova, S. Jeudy, K. Labadie, A. Adrait, M. Lescot, O. Poirot, L. Bertaux, C. Bruley, Y. Coute, E. Rivkina, C. Abergel and J. M. Claverie (2014). "Thirty-thousand-year-old distant relative of giant icosahedral DNA viruses with a pandoravirus morphology." Proc Natl Acad Sci U S A 111(11): 4274-4279.
Lwoff, A. (1957). "The concept of virus." J Gen Microbiol 17(2): 239-253.
Minor, P. D. (2014). "Viruses." Encyclopedia of Life Sciences.
Moreira, D. and P. Lopez-Garcia (2009). "Ten reasons to exclude viruses from the tree of life." Nat Rev Microbiol 7(4): 306-311.
Patrick, F. (2012). "Virocell Concept, The." Encyclopedia of Life Sciences.
Philippe, N., M. Legendre, G. Doutre, Y. Coute, O. Poirot, M. Lescot, D. Arslan, V. Seltzer, L. Bertaux, C. Bruley, J. Garin, J. M. Claverie and C. Abergel (2013). "Pandoraviruses: amoeba viruses with genomes up to 2.5 Mb reaching that of parasitic eukaryotes." Science 341(6143): 281-286.
Raoult, D. (2010). "The post-Darwinist rhizome of life." Lancet 375(9709): 104-105.
Raoult, D., S. Audic, C. Robert, C. Abergel, P. Renesto, H. Ogata, B. La Scola, M. Suzan and J. M. Claverie (2004). "The 1.2-megabase genome sequence of Mimivirus." Science 306(5700): 1344-1350.
Williams, T. A., T. M. Embley and E. Heinz (2011). "Informational gene phylogenies do not support a fourth domain of life for nucleocytoplasmic large DNA viruses." PLoS One 6(6): e21080.
Wu, D., M. Wu, A. Halpern, D. B. Rusch, S. Yooseph, M. Frazier, J. C. Venter and J. A. Eisen (2011). "Stalking the fourth domain in metagenomic data: searching for, discovering, and interpreting novel, deep branches in marker gene phylogenetic trees." PLoS One 6(3): e18011.










1 De Grégoire - 05/06/2015, 15:31
Fascinant ! Un certain Pr. Kerner m'a expliqué il y a longtemps que les virus pouvaient être issus de notre propre organisme. Des cellules renégates en somme, qui se seraient mises à leur compte. C'est ce que tu appelles "vomis parasitiques" ?
Je me demande alors s'il y a un intérêt évolutif à cela, tout comme les entreprises décident d'externaliser des fonctions qu'elles intégraient auparavant. Faciliter l'optimisation du matériel génétique ainsi externalisé, par la reproduction et la mutation rapide des virus créés ? Meilleure communication des gènes "efficaces" entre individus, via ces virus-postier ?
PS : pour une meilleure lisibilité, tes paragraphes mériteraient d'être espacés d'un interligne, puisque tu ne fais pas d'alinéa.
2 De Mathgon - 06/06/2015, 15:50
Encore un bien joli article. Merci!
3 De Lulu Guêpe - 07/06/2015, 09:10
Merci pour l'article, toujours aussi passionnant !
On pourrait peut-être ajouter un résumé de 5 lignes à la fin du texte pour les gens qui n'ont pas tout lu ou pas tout compris, ça pourrait aider :D
Merci pour l'article !
4 De Diti - 07/06/2015, 09:56
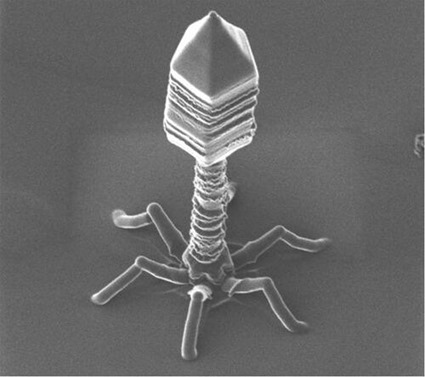
Et je t'ai grillé : la peluche du milieu c'est pas un virus (et essaie pas de dire que c'est un Mimivirus !), je l'ai reconnue c'est la giant competent cell - DH5a E. coli - que Invitrogen (ou Clontech ou encore une autre boîte) refourguait en cadeau!!!!
5 De taupo - 07/06/2015, 17:52
@Grégoire : Oui c'est une hypothèse possible: les virus seraient un agrégat génétique dont l'origine remonterait à une toute petite partie d'un organisme vivant. Il ne faut pas penser cependant qu'il s'agirait là d'une stratéfie de l'organisme initial: c'est très probablement un incident, comme pour les cancerts.
Par contre tu parles d'intérêt évolutif comme si telle ou telle stratégie devait avoir un avantage sur le long terme pour les populations qui l'adoptent: d'une part, uniquement l'effet sur le court terme est sélectionné, d'autre part, sur le long terme, la stratégie de la plupart des virus semble avoir fonctionné puisqu'on les voit partout et en nombre immense.
Pour la lisibilité, ça devrait changer bientôt avec un nouveau design de blog.
6 De taupo - 07/06/2015, 17:58
@Mathgon : Merci beaucoup!
7 De taupo - 07/06/2015, 17:59
@Lulu Guêpe : J'y penserai pour le résumé à la fin, merci pour l'idée et les compliments!
8 De taupo - 07/06/2015, 18:02
@Diti : Pour être parfaitement honnête, il n'y a que deux virus dans cette image. Celle du 'Common Cold' et du 'Flu'. Le reste sont des bactéries. Par contre pas d'E.coli à l'horizon! Celle du milieu c'est Shigella. Tu peux vérifier ici.
9 De Anonymous blog - 07/06/2015, 18:59
[Freaky Friday Parasite] Prodigieux Virus | C@f...
(…)
10 De Anonymous blog - 08/06/2015, 07:50
[Freaky Friday Parasite] Prodigieux Virus - Str...
(…)
11 De Sébastien - 09/06/2015, 08:43
Il y a beaucoup de groupe de recherche qui s'intéressent à la coévolution hôte-pathogène (Surtout chez la Drosophile) et on pourrait imaginer des scénarios évolutifs assez classes. Deux qui me viennent en tête et provoque un rictus malsain :
- On sait que certains virus possèdent des facteurs de traductions (qui permettent à certain, entre autres choses, d'effectuer un décalage du cadre de lecture lors de la traduction afin de produire une seule grosse protéine à partir des deux ORF présents dans leur génome. Protéine qui sera ensuite clivée, blablabla.), on peut imaginer que certains virus possèdent des protéines permettant de faire du RNA editing pour détourner des ARNm (Ou non messagers d'ailleurs) de l'hôte et les utiliser comme protéine de capside, ou autre. Typiquement, l'introduction d'un codon start alternatif avec un cadre de lecture décalé ou des changements de bases par une adénosine déaminase agissant sur l'ARN d'origine virale.
- L'infection chronique de toute une population d'une espèce par un virus. Au hasard d'une infection, il récupère un gène de l'hôte (Par contre, je ne suis pas sûr que ça puisse se faire sans avoir un impact sur le contenu génique du virus initial) qui, lorsqu'il est transféré par transduction à un autre individu lui procure un avantage sélectif. Le virus subit une sélection positive dans la population et la présence de cette seconde copie d'origine virale diminue la pression de sélection sur la copie présente chez l'organisme. Le hasard de la reproduction donne des individus dépourvus du gène en question, ce qui rend l'infection obligatoire par le virus pour survivre/avoir une fitness convenable. Je sais pas si c'est super clair mais je trouve ça drôle (Bien sûr cette situation ne serait que temporaire.)
12 De Anonymous blog - 29/06/2015, 00:43
CRISPR, la mutagenèse qui croustille
(…)
13 De Anonymous blog - 02/05/2016, 07:48
[Freaky Friday Parasite] Prodigieux Virus - Str...
(…)
14 De Dirtdiver60 - 20/07/2016, 18:21
Ou là ! Tu me prends aux sentiments avec les virus dis donc ! Moi qui suis pro-microbiologiste :D <3 (PS : moi aussi j'ai une peluche giant microbes :D on s'en est offerte une à chacune avec ma meilleure amie de la fac pour l'obtention de notre bac +2 en microbio : le neurone avec le chapeau de diplômé xD)